次世代シーケンサーデータ解析 short read micro re-aligner [げのむ]
次世代シーケンサーのデータをConsedで扱うのに奮闘していたのが去年の秋
もうすぐ1年経つのだな
Davidの話だと次のConsed20ではデータ構造もphdballから一気に変更して次世代完全対応を狙っているようだが、現時点で全ゲノム解析ではConsed19では対応は不可能だ。
ということでどれを使っているかというと、結局無難なbwa
Consedの次にMaq、bowtieと試したがとりあえずはbwa/bowtie/novoの3つで変異の抽出までやってからフィルターをかけることで落ち着いた。
構造解析ではガッツリ厳しいパラメーターで、変異探索はゆるめの2回が基本になりそう。
1000genomesもMaqからbwaに落ち着いたようなので当面はbwaかな
ということで
bwa/bowtie/novo --> samtools --> SIFT
で目的は果たしているが、苦戦しているのがindelのローカルアライメント
変異よりもgapのペナルティーのほうが厳しくせざるを得ないのでshort readのアライメントはindelが厳しい
そこでMultiple Sequence Alignment (MSA) を狭い領域でやる手法があるのだが、これに苦戦
PicardもGenome Analysis Toolkitも一通り立ち上げたが、どうもおかしい
samtoolsよりもGATKのほうがフォーマットに厳しく、特にbwaの0処理のバグを許さない
そこでbwaのアライメントのバグデータを取り除いてsamの状態でsortしてからbamに変換
しかも一旦sortしてもGATK IntervalCleanerが名前順にsortするので、結局samtoolsでまたsortのやり直し
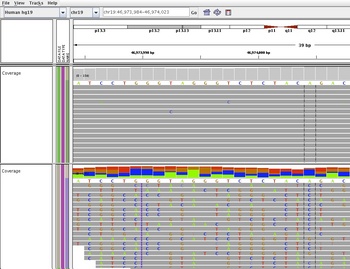
そこまで苦労しても出てくるのが上の図、明らかにlocal alignment がうまくいっていない
どうも、どこかの落とし穴にはまっているらしいが、情報が少ないので解決できず
hg19が問題なのかインデックスが問題なのか不明
一方でSRMAもbwa/samtoolsで出てきたファイルを許容しないので、ゴリゴリ汚いデータを処理してからかけるがどうも上手くない
結局32GBのメモリ程度だと全ゲノムに対応しきれないらしい。1番染色体の途中でheap errorで落ちる
領域ごとにぶった切って処理して回収という案も考えたが、ルーチン化するには処理が面倒なので、こちらも頓挫
土日もがんばったのに結局うまくいかず
しょげるなぁ
もうすぐ1年経つのだな

Davidの話だと次のConsed20ではデータ構造もphdballから一気に変更して次世代完全対応を狙っているようだが、現時点で全ゲノム解析ではConsed19では対応は不可能だ。
ということでどれを使っているかというと、結局無難なbwa
Consedの次にMaq、bowtieと試したがとりあえずはbwa/bowtie/novoの3つで変異の抽出までやってからフィルターをかけることで落ち着いた。
構造解析ではガッツリ厳しいパラメーターで、変異探索はゆるめの2回が基本になりそう。
1000genomesもMaqからbwaに落ち着いたようなので当面はbwaかな
ということで
bwa/bowtie/novo --> samtools --> SIFT
で目的は果たしているが、苦戦しているのがindelのローカルアライメント
変異よりもgapのペナルティーのほうが厳しくせざるを得ないのでshort readのアライメントはindelが厳しい
そこでMultiple Sequence Alignment (MSA) を狭い領域でやる手法があるのだが、これに苦戦
PicardもGenome Analysis Toolkitも一通り立ち上げたが、どうもおかしい
samtoolsよりもGATKのほうがフォーマットに厳しく、特にbwaの0処理のバグを許さない
そこでbwaのアライメントのバグデータを取り除いてsamの状態でsortしてからbamに変換
しかも一旦sortしてもGATK IntervalCleanerが名前順にsortするので、結局samtoolsでまたsortのやり直し
そこまで苦労しても出てくるのが上の図、明らかにlocal alignment がうまくいっていない
どうも、どこかの落とし穴にはまっているらしいが、情報が少ないので解決できず
hg19が問題なのかインデックスが問題なのか不明
一方でSRMAもbwa/samtoolsで出てきたファイルを許容しないので、ゴリゴリ汚いデータを処理してからかけるがどうも上手くない
結局32GBのメモリ程度だと全ゲノムに対応しきれないらしい。1番染色体の途中でheap errorで落ちる
領域ごとにぶった切って処理して回収という案も考えたが、ルーチン化するには処理が面倒なので、こちらも頓挫
土日もがんばったのに結局うまくいかず
しょげるなぁ



相変わらず意味が分からない日本語で、すごい。
by molihua (2010-09-14 12:51)
しょげんなよぅ。 (・∀・)
by コバ (2010-09-14 22:51)
おっとさんこんばんはー。
おばかのゆきぽんです。おばかです。
consedとか懐かしい響きですねぇ。
最近sequenceしていないのでやり方忘れそうです。
と言うか忘れてますが…。
ゆきぽん結婚したのですが、仕事で無理しすぎてまたまた体壊してもうてえへへ…。
今月一杯で辞める事にしますた。
だからまた暫くは研究とはおさらばになるけど、ココに勉強しに来ます。
解析はさっっっっぱり分からないんだけども。
ハニーさんとちびハニーさんにおよろしく。
ハニーさんの方にコメするのは勇気がいるのです…
by ゆきぽん (2010-09-21 18:59)
>ゆきぽん
ごめんね、アホなblogで(ノ∀`)
by はにぃ (2010-09-26 23:14)
>はにぃさん
こんばんはー!はにぃさん、こんばんわー!
毎回楽しく読ませてもらっています。
違いますよ、コメントが凄い沢山で、
その中にぽちっと書いて良いのかちょいと気後れしちゃうんです。
はにぃさんみたいな素敵な奥さんとお母さんになれる様に
私も頑張ります!
おっとさんには無理だと言われそうですけどね。
ふへぇー。
by ゆきぽん (2010-09-27 02:09)
>ゆきぽむ
おひさです。忙しいです。へろへろです。
今度遊びにきてねー
by カオナシ (2010-10-25 23:29)